tRNA
Den korte formen tRNA står for overføring av RNA . Overførings-RNA er korte ribonukleinsyre-tråder (RNA) som spiller en viktig rolle i dannelsen av proteiner ( proteinbiosyntese ) i hver celle ved å overføre individuelle aminosyremolekyler. Lengden på modne tRNA-tråder er vanligvis mellom 73 og 95 nukleotider . Ved å bruke basistripletten av antikodonet deres , overfører de riktig aminosyre i hvert tilfelle i henhold til den genetiske koden for et kodon på mRNA under translasjon på ribosomet .
struktur

Pu : purinbase
Py : pyriminbase
T : tymidin
Ψ : pseudouridin
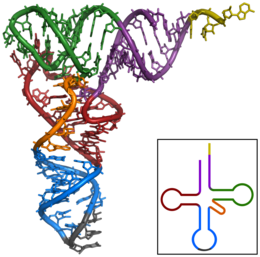
Som regel består tRNA-molekyler av 73 til 95 nukleotider av en enkelt RNA-streng. I tillegg til de fire grunnleggende byggesteinene i RNA ( adenosin , uridin , cytidin og guanosin ) inneholder tRNA en rekke forskjellige modifiserte standardbaser . For eksempel har man nukleosidene dihydrouridin (D), inosin (I), 2-tiouridin (s 2 U), 4-tiouridin (s 4 U), pseudouridin (Ψ), N 4- acetylcytidin (ac 4 C) og 5-metyluridin ( tymidin ) (T) identifisert.
Parring av konjugerende nukleobaser forekommer i hvert tRNA-molekyl, og danner dermed dobbeltstrengede regioner. I en todimensjonal representasjon har tRNA en kløverbladlignende sekundærstruktur . En stamme og tre løkker kan skilles fra denne strukturen: en dihydrouridin , en TΨC og en antikodonsløyfe (se figur).
- D-arm (dihydrouridin-arm): Den såkalte dihydrouridin-sløyfen på enden av D-armen skylder navnet sitt på dihydrouracilrester som den ofte inneholder . Det vises vanligvis til venstre i figurer. Imidlertid inneholder en dihydrouridinarm ikke alltid de eponyme dihydrouracilrester. Hovedformålet med dihydrouridin-sløyfen er å gjenkjenne tRNA ved aminoacyl-tRNA-syntetase.
- Lav antikodon: På antikodonsløyfen er det en spesifikk basetriplett , den såkalte antikodon . Sløyfen består alltid av fem konjugerende par og syv uparede baser, hvorav tre danner antikodonet. Dette samhandler med det komplementære kodonet til mRNA som skal leses (se avsnitt Funksjon).
- T-sløyfe (TΨC-sløyfe): TΨC-sløyfen vist på figurene til høyre inneholder syv uparrede baser, hvorav tre danner den samme navnesekvensen TΨC. T står for (ribo-) tymidin, Ψ for pseudouridin og C for cytidin.
- Variabel sløyfe: Den såkalte variable sløyfen ligger mellom TΨC-armen og antikodonarmen; dette varierer i lengde avhengig av tRNA.
- Akseptorstamme: Akseptorstammen fosforyleres i 5'-enden. Dens 3'-ende viser alltid sekvensen CCA-3 '. Den adenosin (A) ved 3'-enden presenterer sin ribose , som spiller en viktig rolle. Fordi 3'-OH-gruppen i ribosen, karboksygruppen til en tilsvarende aminosyre, i denne enden blir forestret og dermed bundet til tRNA (se avsnitt Funksjon).
De modifiserte basene er hyppigst funnet blant de ikke-konjugerte basene i trefoil-strukturen. Den faktiske tredimensjonale tertiære strukturen er mer som en "L" -form, hvor de to utstående delene er dannet av aminosyreakseptorstammen og antikodonsløyfen. Disse funksjonelt viktige regionene er derfor så langt fra hverandre som mulig. Den tredimensjonale strukturen til tRNA fra gjær ble allerede belyst i 1974 med en oppløsning på 300 pm (3 Å ) uavhengig av to grupper ledet av Rich og Klug. I 2000 ble en struktur med en forbedret oppløsning på 1,93 Å publisert.
En tRNA Ala fra S. cerevisiae
funksjon
Under translasjon i proteinbiosyntese må den passende aminosyren være festet til peptidkjeden i ribosomet i samsvar med den genetiske koden for hver basetriplett på mRNA. Denne oppgaven formidles av tRNA. Det er minst en, men ofte flere, forskjellige tRNAer for hver aminosyre.
Lasting av tRNA med en aminosyre
Ved ATP- forbruk lastes tRNAer spesifikt med den assosierte aminosyren i 3'-enden av den respektive aminoacyl-tRNA-syntetase, avhengig av sekvens . For dette formål er karboksygruppen i aminosyren bundet i en esterbinding til 3'- hydroksylgruppen i ribosen i adenosinet, slik at en aminoacylgruppe blir dannet. Aminoacyl-tRNA-syntetasen gjenkjenner ofte antikodonet på tRNA for dette formålet. Imidlertid kan andre strukturelle elementer også spille en rolle i anerkjennelsen, hovedsakelig akseptorstammen.
Et spesielt tilfelle er tRNAene som er lastet med alanin (tRNA Ala ). Uavhengig av organismen har tRNA Ala et G-U basepar i posisjonene 3 og 70 i akseptorstammen. Hvis en av disse to basene byttes mot en annen ved (målrettet) mutagenese, kan den resulterende tRNA ikke lenger fylles med alanin av alanyl-tRNA Ala- syntetasen.
Opprinnelig ble det antatt at det er nøyaktig en aminoacyl-tRNA-synthetase for hver aminosyre i alle organismer, som kan laste alle tRNAer som tilhører den tilsvarende aminosyren. Det ble imidlertid senere oppdaget at noen organismer mangler en eller flere aminoacyl-tRNA-syntetaser, men at de fremdeles kan inkorporere de tilsvarende aminosyrene i proteinene sine og også ha de nødvendige tRNAene. Disse må derfor ha en annen mekanisme for lasting av disse tRNAene. Mange arkeaer, bakterier, kloroplaster og mitokondrier mangler en aminoacyl-tRNA-syntetase for glutamin . I stedet binder aminoacyl-tRNA-syntetasen for glutamat den til både tRNA for glutamat og tRNA for glutamin. Det "feil" bundne glutamatet til tRNA for glutamin blir deretter omdannet til glutamin av en transamidase (Glu-tRNA Gln amidotransferase).
Inkorporering av aminosyrer i den begynnende kjeden
De aminoacylerte tRNAene brukes av ribosomene for proteinsyntese. Hvis antikodonet samsvarer med den tilsvarende basekodonen til messenger RNA (mRNA) , kan tRNA feste seg der og feste den transporterte aminosyren til det resulterende proteinet .
I følge den genetiske koden , ville et tRNA måtte eksistere for hver basetriplett som koder en aminosyre og ikke er en stoppkodon - vanligvis 61. Det ble imidlertid funnet at antall tRNAer avviker betydelig nedover. Det nøyaktige antallet er forskjellig i organismer, men det er ikke mindre enn 31 og ikke mer enn 41. Ikke desto mindre brukes i alle organismer alle basetripletter til proteinkoding. Dette avviket forklares med wobble-teorien .
| Antikodonbase (tRNA) | Kodonbase (mRNA) |
|---|---|
| C. | G |
| EN. | U |
| G | U eller C |
| JEG. | U, C eller A. |
| U | A eller G; i kloroplaster og mitokondrier også U eller C |
Denne teorien, som går tilbake til Francis Crick , sier at de to første basene i kodonet og de to siste basene i antikodonet danner hydrogenbindinger i henhold til "klassisk" Watson-Crick baseparing; G pares alltid med C og A med U. I følge Cricks undersøkelser kan muligheten for baseparring mellom den tredje basen til kodonen og den første basen til antikodonet utvides og følger ikke denne strenge regelen. Man snakker også om "wobble position".
For eksempel, hvis den første basen til antikodonet er en U, kan den gjenkjenne et A eller G av kodonet. Noen ganger er denne første basen også en inosin, den kan samhandle med både U, C og A på kodonen. Resultatene er oppsummert i tabellen til høyre.
Den første basen av antikodonet blir ofte modifisert. Dette påvirker gjenkjennelsen av den tilsvarende tredje basen av kodonen. For eksempel, hvis den første basen av antikodonet er et lysidin (k 2 C), gjenkjennes dette bare av en A i bakterier. I mitokondriene til noen eukaryoter er 5-formylcytidin (f 5 C) baseparret med A eller G. I pighuder og mitokondrier av blekksprut er det blitt oppdaget at 7-metylguanosin (m 7 G) kan samhandle med alle fire baser.
På grunn av det faktum at den genetiske koden er en degenerert kode der flere basetripletter kan kode for den samme aminosyren, er ofte bare to baser nødvendige for gjenkjenning, siden basetripletter for den samme aminosyren ofte bare skiller seg i en base .
Initiativtaker
I syntesen av et polypeptid stiller startprosessen ( initiering ) andre krav til tRNA enn forlengelsen ( forlengelsen ) av kjeden. Siden syntesen vanligvis starter ved et kodon AUG (koding for metionin), krever cellen en spesiell tRNA egnet for initiering for startkodonet AUG. Denne tRNA i , også kalt initiator tRNA, skiller seg fra en annen metioninspesifikk tRNA Met , som er nødvendig for gjenkjenning av AUG-kodonet i en leseramme. Selv om begge tRNAene er fylt med metionin av samme metionyl-tRNA-syntetase, kan ikke tRNA Met brukes til startkodonet, bare tRNA i Met .
I bakterier, for eksempel E. coli , blir initiatoren tRNA gitt som tRNA i fMet . “F” i navnet indikerer at tRNA fylt med metionin er modifisert med en formylrest . Denne reaksjon katalyseres ved hjelp av en metionyl tRNA-formyl-transferase ( EC 2.1.2.9 ), hvor N 10 formyl-tetrahydrofolat (10-CHO-THF) omdannet til tetrahydrofolsyre (THF). TRNA i fMet skiller seg på viktige punkter fra tRNA Met , slik at sistnevnte ikke blir gjenkjent av formyltransferase og derfor bare tRNA i fMet brukes for initieringen. Så det er ingen baseparring mellom det første cytosinet og adeninet i akseptorstammen (se bilde). Det er tre påfølgende G - C basepar i antikodonstammen. Til slutt har initiatoren tRNA i D-sløyfen en CCU-sekvens. Sammen skaper disse forskjellene en litt annen konformasjon.
Initiatoren tRNA i eukaryoter, så vel som i archaea, er ikke formylert. Etter translasjon kan det dannede proteinet modifiseres. I bakterier, for eksempel, blir den N-terminale formylresten vanligvis delt av . Metioninet ved N-terminalen av polypeptidet kan da også deles av, det samme kan eukaryoter og archaea.
Biosyntese av tRNA
tRNA-gener
Organismer varierer i antall tRNA- gener på genomet. De nematode Caenorhabditis elegans , en typisk modellorganisme for genetiske studier, har 29,647 gener i sin kjerne genom, 620 av hvilke koder for tRNA. Den bakegjær har 275 tRNA-gener i deres genom .
I prokaryoter er flere gener vanligvis gruppert i operoner . Genene kan kode for proteinsekvenser eller forskjellige RNA-produkter, inkludert tRNA. Som regel er flere tRNAer kombinert i en operon, og disse kan også inneholde proteinkodende gener.
I eukaryoter må det skilles mellom tRNA-gener på DNA i cellekjernen og tRNA-gener på DNA fra mitokondrier (så vel som DNA-holdige hydrogenosomer ) eller plastider som kloroplaster . I plastider er genene organisert i operoner som ligner prokaryoter (se endosymbiont teori ). Utstyret med tRNA-gener (f.eks. 30 tRNA-gener i Marchantia polymorpha ) inkluderer alle tRNA-grupper som er nødvendige for proteinsyntese i plastider.
Imidlertid er et komplett sett med tRNA-gener ikke alltid tilgjengelig i mitokondrier, slik at - avhengig av arten - tRNA må importeres fra cytosolen til organellen. Mens i nesten alle opisthoconta (f.eks. Hos mennesker eller gjær ) blir alle mitokondrielle tRNA-gener kodet av mitokondrie-DNA, for eksempel hos cnidarians eller apikomplekser , må de fleste tRNAer importeres.
Transkripsjon av tRNA
Prokaryoter har bare en RNA-polymerase . Dette transkriberer leserammen til en operon, slik at et polycistronic mRNA vanligvis produseres. Om nødvendig må tRNAene ekstraheres fra dette ved endonukleolytiske kutt i de intercistroniske områdene.
I eukaryoter transkriberes tRNA i kjernen av RNA-polymerase III. I motsetning til dette transkriberes proteinkodende gener der av RNA-polymerase II. TRNA i plastider transkriberes av en RNA-polymerase som ligner på prokaryoter (se også teori om endosymbiont ). Denne RNA-polymerasen er også kodet i plastidgenomet, mens en annen må importeres fra cytoplasmaet . Det er to RNA-polymeraser i mitokondriene, som begge må importeres. De er relatert til det kjernekodede plastidenzymet.
I archaea , som i prokaryoter, er det bare en RNA-polymerase for transkripsjon. Sammenlignet med prokaryoter er dette imidlertid mer komplekst. Størrelsen, antall underenheter og deres aminosyresekvenser er mer sammenlignbare med eukaryote RNA-polymeraser.
Modning av tRNA-forløperne
Pre-tRNAene som følge av transkripsjonen kan inneholde introner . I bakterier , det skjøting av disse introner foregår autokatalytisk , mens intronene i eukaryoter og archaea blir fjernet ved spleising tRNA endonuklease .
I eukaryoter finner både tRNA-syntese og den første prosessen etter transkripsjon, slik som trimming av 5'- og 3'-endene, basemodifikasjoner og posttranskripsjonell tilsetning av 3'CCA-sekvensen sted i kjernen. Eksporten til cytosolen formidles av proteiner som Los1. I cytosolen skjer spleising på den ytre membranen av mitokondriene, hvis introner er tilstede, og også ytterligere modifikasjoner til tRNA har modnet.
Dannelse av tRNAene
Den øvre halvdelen av tRNA (fra D-armen og akseptorstammen med den 5'-terminerende fosfatenheten og den 3'-avsluttende CCA-gruppen) og den nedre halvdelen (fra T-armen og antikodonarmen) er enheter som er uavhengige i struktur og funksjon. Det antas at den øvre halvdelen utviklet seg først, med CCA-gruppen opprinnelig - det vil si i en tidlig RNA-verden - kan ha merket tRNA-lignende molekyler for replikasjonsformål (en slik markør kalles 'Genomic Tag' på engelsk) ). Den nedre halvdelen kunne blitt opprettet senere som en utvidelse, f.eks. B. da proteinbiosyntesen startet og RNA-verdenen endret seg til en ribonukleoproteinverden ( RNP-verden ). Et slikt scenario kalles 'Genomic Tag Hypothesis' på engelsk. Faktisk spiller tRNA-lignende molekyler fremdeles en viktig katalytisk rolle (ergo som ribozymer ) i replikasjon. De kan sees på som molekylære (eller kjemiske) fossiler som, i likhet med " levende fossiler ", gir ledetråder om evolusjonens tidlige forløp.
Variasjoner av tRNA dannes i dyreriket
Den "klassiske" formen av tRNA vist ovenfor med en trearmet "kløverbladstruktur" er bekreftet i tidligere undersøkelser for de fleste tRNAer. I noen stammer ble det imidlertid funnet kraftig modifiserte mitokondrie-tRNAer (mt-tRNAer) der forskjellige deler av den normale strukturen ble sterkt modifisert eller fullstendig tapt. Det virker usannsynlig at dette er pseudogener som har blitt funksjonsløse på grunn av mutasjoner . Når det gjelder en tRNA-sekvens, mangler D-armen i hele dyreriket, noe som antyder at denne sekvensen allerede var modifisert i forfedrene til alle moderne dyr. Spesielt høyt modifiserte mitokondrie-tRNA-strukturer er identifisert i nematoder og arachnids . Noen mt-tRNAer har mistet store deler av sine vanlige strukturer uten å miste funksjonaliteten. Vanligvis, i stedet for den tapte strukturen, kan en erstatningsstruktur, f.eks. B. en ekstra sløyfe (TV-erstatningssløyfe) dannes. Det er ennå ikke avklart hvorfor tapet av en ellers svært konservert struktur, hvis mutasjon nesten alltid skal være dødelig i henhold til alle antagelser, er mulig her som et unntak.
Se også
litteratur
- Reginald Garrett, Charles M. Grisham: Biokjemi. (International Student Edition). 4. utgave. Cengage Learning Services, 2009, ISBN 978-0-495-11464-2 .
- Peter Karlson, Detlef Doenecke, Jan Koolman, Georg Fuchs, Wolfgang Gerok: Karlsons Biochemie und Pathobiochemie . 15. utgave. Georg Thieme, 2005, ISBN 3-13-357815-4 , s. 142.
- Dieter Soll (red.), Uttam L. RajBhandary (red.), TL Rajbhandary: tRNA: struktur, biosyntese og funksjon . Asm Press, 1995, ISBN 1-55581-073-X .
- EM Phizicky, AK Hopper: tRNA-biologi lades foran. I: Genes Dev . 24 (17), 2010, s. 1832-1860. PMID 20810645 ; PMC 2932967 (gratis fulltekst, PDF).
- MA Rubio, AK Hopper: Overførings-RNA beveger seg fra cytoplasma til organeller. I: Wiley Interdiscip Rev RNA . 2 (6), 2011, s. 802-817. PMID 21976284 ; doi: 10.1002 / wrna.93
- Paul Schimmel: Den nye kompleksiteten i tRNA-verdenen: pattedyrs tRNAs utover proteinsyntese . I: Naturanmeldelser. Molekylær cellebiologi . 6. september 2017, doi : 10.1038 / nrm.2017.77 , PMID 28875994 .
- Kunal Chatterjee, Regina T. Nostramo, Yao Wan, Anita K. Hopper: tRNA-dynamikk mellom kjernen, cytoplasma og mitokondrieoverflate: Beliggenhet, beliggenhet, beliggenhet . I: Biochimica Et Biophysica Acta . 27. november 2017, doi : 10.1016 / j.bbagrm.2017.11.007 , PMID 29191733 .
Individuelle bevis
- ↑ RK Kumar, DR Davis: Syntese og studier av effekten av 2-tiouridin og 4-tiouridin på sukkerkonformasjon og RNA-dupleksstabilitet. I: Nucleic Acids Research . Volum 25, nummer 6, mars 1997, s. 1272-1280. PMID 9092639 , PMC 146581 (fri fulltekst).
- ↑ Yuchen Liu et al.: Biosyntese av 4-tiouridin i tRNA i den metanogene arkeon Methanococcus maripaludis. I: Journal of Biological Chemistry . 287, 17. august 2012, s. 36683-36692, doi: 10.1074 / jbc.M112.405688
- ^ H. Grosjean, R. Gupta, ES Maxwell: Modifiserte nukleotider i arkeiske RNA. I: P. Blum (red.): Archaea: nye modeller for prokaryotisk biologi. Caister Academic Press, Norfolk, UK 2008, s. 171-196.
- ↑ SH Kim et al. Tredimensjonal tertiære strukturen til gjær fenylalanin overføring RNA. I: Vitenskap . 185 (149), 1974, s. 435-440. PMID 4601792 ; doi: 10.1126 / science.185.4149.435 .
- ↑ JD Robertus et al.: Struktur av gjærfenylalanin tRNA ved 3 Å oppløsning. I: Natur . 250 (467), 1974, s. 546-551. PMID 4602655 ; doi: 10.1038 / 250546a0 .
- ↑ a b H. Shi, PB Moore : Krystallstrukturen til gjærfenylalanin tRNA ved 1,93 Å oppløsning: en klassisk struktur revidert. I: RNA . 6 (8), 2000, s. 1091-1105. PMID 10943889 ; PMC 1369984 (gratis fulltekst, PDF).
- ^ Reginald Garrett, Charles M. Grisham: Biokjemi. (International Student Edition). 4. utgave. Cengage Learning Services, 2009, ISBN 978-0-495-11464-2 , s. 958.
- ↑ K. Horiuchi, M. Harpel, L. Shen, Y. Luo, K. Rogers, R. Copeland: Mekanistiske Studier av Reaksjon Kobling i Glu-tRNAGln Amidotransferase. I: Biokjemi . Volum 40, nr. 21, mai 2001, s. 6450-6457, doi: 10.1021 / bi002599l .
- ↑ Katharina Munk (red.): Pocket lærebok Biology: Genetics. 1. utgave. Thieme Verlag, 2010, ISBN 978-3-13-144871-2 , s. 188.
- ^ Peter Karlson, Detlef Doenecke, Jan Koolman, Georg Fuchs, Wolfgang Gerok: Karlsons Biochemie und Pathobiochemie. 15. utgave. Thieme, 2005, s. 142.
- ^ F. Crick: Opprinnelsen til den genetiske koden . I: J Mol Biol . teip 38 , nr. 3 , 1968, s. 367-379 , doi : 10.1016 / 0022-2836 (68) 90392-6 , PMID 4887876 .
-
↑ H. Lodish et al.: Molecular Biology of the Cell. 5. utgave. WH Freeman, New York 2004, ISBN 0-7167-3136-3 . (Utdrag) (engelsk)
Harvey Lodish et al.: Molecular Cell Biology. Spectrum Academic Publishing House, 2001, ISBN 3-8274-1077-0 . - ↑ a b A. Ambrogelly et al.: Naturlig utvidelse av den genetiske koden. I: Nature Chemical Biology . 3 (1), 2007, s. 29-35. PMID 17173027 ; doi: 10.1038 / nchembio847 .
- ^ Reginald Garrett, Charles M. Grisham: Biokjemi. (International Student Edition). 4. utgave. Cengage Learning Services, 2009, ISBN 978-0-495-11464-2 , s. 959.
- ^ Reginald Garrett, Charles M. Grisham: Biokjemi . (International Student Edition). 4. utgave. Cengage Learning Services, 2009, ISBN 978-0-495-11464-2 , s. 977.
- ^ Reginald Garrett, Charles M. Grisham: Biokjemi . (International Student Edition). 4. utgave. Cengage Learning Services, 2009, ISBN 978-0-495-11464-2 , s. 966.
- ↑ Donald Voet, Judith G. Voet: Biokjemi. Wiley-VCH, 1994, ISBN 3-527-29249-7 , s. 928.
- ↑ SE Kolitz, JR Lorsch: Eukaryot initiativtaker tRNA: finjustert og klar til handling. I: FEBS Letters . 584 (2), 21. januar 2010, s. 396-404 ( doi: 10.1016 / j.febslet.2009.11.047 , PMC 2795131 (fri fulltekst). PMID 19925799 ).
- ↑ WormBase : Versjonsmerknader - WS187 . 25. januar 2008.
- Sp J. Spieth, D. Lawson: Oversikt over genstruktur. I: WormBook . 2006, s. 1-10. PMID 18023127 ; PDF (gratis fullteksttilgang)
- ↑ LH Hartwell et al.: Genetikk: Fra gener til genomer. 2. utgave. McGraw-Hill, New York 2004, s.264.
- ^ André Schneider: Mitokondriell tRNA-import og dens konsekvenser for mitokondriell oversettelse. I: Annu Rev Biochem . 80, 2011, s. 1033-1053. PMID 21417719 ; doi: 10.1146 / annurev-biochem-060109-092838
- ↑ RJ White: Regulering av RNA-polymeraser I og III av retinoblastomproteinet: en mekanisme for vekstkontroll? I: Trender i biokjemiske vitenskaper . 22 (3), 1997, s. 77-80. PMID 9066256 ; doi: 10.1016 / S0968-0004 (96) 10067-0
- ↑ J. Abelson, CR Trotta, H. Li: tRNA Splicing. I: J Biol Chem 273 (21), 1998, s. 12685-12688. PMID 9582290 ; PDF (gratis fullteksttilgang)
- ↑ AK Hopper, DA Pai, DR Engelke: Cellular dynamikk tRNA og deres gener. I: FEBS Lett . 584 (2), 2010, s. 310-317. PMID 19931532 ; PMC 2818515 (gratis fulltekst, PDF).
- ^ Nancy Maizels, Alan M. Weiner: The Genomic Tag Hypothesis - What Molecular Fossils Tell Us about the Evolution of tRNA. I: RNA-verdenen. 2. utgave. Cold Spring Harbour Laboratory Press, 1999, ISBN 0-87969-561-7 . (PDF)
- ↑ DR Wolstenholme: Dyr mitokondrie DNA: struktur og evolusjon. I: DR Wolstenholme, KW Jeon (red.): Mitokondrielle genomer. Academic Press, New York 1992, s. 173-216.
- ^ Y. Watanabe et al.: Primære og høyere ordensstrukturer av nematode (Ascaris suum) mitokondrie-tRNAer som mangler enten T- eller D-stammen. I: Journal of Biological Chemistry . 269 (36), s. 22902-22906. PMID 8077242 ; PDF (gratis fullteksttilgang)
- ↑ Susan E. Masta, Jeffrey L. Boore: Parallell evolusjon av avkuttede overførings-RNA-gener i Arachnid Mitochondrial Genomes. I: Molecular Biology and Evolution . 25 (5), 2008, s. 949-959. PMID 18296699 ; PDF (gratis fullteksttilgang)
weblenker
- RNA-modifikasjonsdatabasen ( Memento fra 7. april 2013 i Internet Archive ) - Omfattende database om modifiserte baser i tRNA
- Database over pattedyrs mitokondrie-tRNAer
- Den genomiske tRNA-databasen (GtRDB)
- Alexandra Kühnlein et al. : tRNA-sekvenser kan settes sammen i en replikator , i: eLife 10 2. mars 2021, e63431, doi: 10.7554 / eLife.63431 . Til:
- Livets opprinnelse: Kylling-og-egg-problemet , den: ScienceDaily fra 2. mars 2021








